NGS 데이터 분석에서는 데이터의 신뢰성과 정확도를 평가하는 것이 매우 중요하다. 그중에서도 PHRED 품질 점수(Phred Quality Score)는 각 염기 서열의 신뢰도를 수치화하여 제공하며, 고품질 데이터 확보와 데이터 해석의 신뢰성을 높이는 데 큰 역할을 한다. 이번 글에서는 PHRED 품질 점수가 무엇인지, 왜 중요한지, 그리고 파이썬 코드로 FASTQ 데이터의 품질 점수를 확인하는 방법까지 설명하겠다.
PHRED Quality Score란?
PHRED 품질 점수는 NGS(Next-Generation Sequencing) 데이터를 처리할 때 각 염기 서열의 정확도를 나타내는 지표이다. 이 점수는 각 염기의 검출 오류 확률을 수치화하여 표현하며, 데이터의 신뢰도를 높이기 위해 중요한 역할을 한다. PHRED 품질 점수는 다음과 같은 수식으로 계산된다:
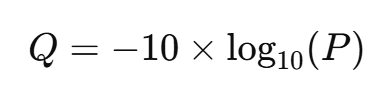
- Q: 품질 점수
- P: 염기가 잘못 검출될 확률
즉, Q 값이 높을수록 염기 검출의 정확도가 높고, 오류 확률이 낮다는 것을 의미한다. 예를 들어, Q20은 오류 확률이 1%, Q30은 0.1% 오류 확률을 나타낸다. 이는 데이터의 신뢰도를 높이기 위한 지표로써, 연구의 정확도를 높이는 데 매우 중요한 역할을 한다.
왜 PHRED 품질 점수를 확인해야 하는가?
- 데이터의 신뢰성 확보: 품질 점수는 각 염기가 얼마나 정확하게 검출되었는지를 나타내므로, 분석에 앞서 데이터의 신뢰성을 평가할 수 있다.
- 비용과 시간의 효율성: 잘못된 염기 데이터로 분석을 진행하면 불필요한 비용과 시간을 낭비하게 되며, 결과의 정확도도 떨어질 수 있다.
- 변이 검출의 정확성: 암 연구나 유전자 변이 분석에서는 미스매치가 변이인지 기술적 오류인지를 구분하는 것이 중요하다. 품질 점수가 낮은 위치에서 발생한 미스매치는 오류일 가능성이 높아, 이를 걸러내는 데 품질 점수가 필요하다.
PHRED 품질 점수의 아스키코드화 이유와 해석 방법
FASTQ 파일에서는 각 염기의 품질 점수가 아스키(ASCII) 코드 문자로 저장된다. 이는 파일의 저장 공간을 줄이고 데이터 처리를 간편하게 하기 위해서이다. FASTQ 파일의 데이터는 보통 수백만 개의 염기로 구성되기 때문에, 각 염기의 품질 점수를 숫자로 직접 기록하면 파일 크기가 지나치게 커지게 된다. ASCII 문자를 활용하면 숫자 대신 문자로 품질 점수를 기록하여 데이터 압축 효과를 높일 수 있다.
- 품질 점수와 아스키 문자 관계: 각 문자는 고유의 아스키 코드 값을 가지며, 품질 점수와 대응된다. 일반적으로 33을 더하거나(Sanger 포맷), 64를 더한 값으로 변환된 문자가 기록된다.
- 예를 들어, 품질 점수가 30일 때 Sanger 포맷에서는 ASCII !부터 시작하여 30 + 33 = 63, 즉 문자 ?로 저장된다.
- 해석 방법: 품질 점수를 얻기 위해 각 문자의 아스키 값에서 33(Sanger 포맷)이나 64(Illumina 1.3+ 포맷)을 빼면 실제 품질 점수를 확인할 수 있다.
예시 코드를 통해 FASTQ 파일에서 품질 점수를 추출하고 해석하는 방법을 살펴보겠다.
파이썬 코드로 FASTQ 데이터의 품질 점수 분석하기
FASTQ 파일의 품질 점수를 파싱하여 평균, 중위값, 상위 25%, 하위 25%를 계산하는 파이썬 코드를 작성해 보겠다. 이 코드에서는 Bio.SeqIO 모듈을 사용하여 FASTQ 파일을 읽고, 각 염기의 품질 점수를 추출하여 계산한다.
from Bio import SeqIO
import numpy as np
# FASTQ 파일 경로 설정
fastq_file = "your_fastq_file.fastq"
# 품질 점수를 저장할 리스트 생성
quality_scores = []
# FASTQ 파일 읽기 및 품질 점수 추출
for record in SeqIO.parse(fastq_file, "fastq"):
quality_scores.extend(record.letter_annotations["phred_quality"])
# 품질 점수의 평균, 중위값, 상위 25%, 하위 25% 계산
mean_quality = np.mean(quality_scores)
median_quality = np.median(quality_scores)
upper_25th = np.percentile(quality_scores, 75)
lower_25th = np.percentile(quality_scores, 25)
# 결과 출력
print(f"품질 점수 평균: {mean_quality:.2f}")
print(f"품질 점수 중위값: {median_quality:.2f}")
print(f"상위 25% 품질 점수: {upper_25th:.2f}")
print(f"하위 25% 품질 점수: {lower_25th:.2f}")이 코드에서는 품질 점수를 저장할 리스트를 생성한 뒤 FASTQ 파일을 읽어 각 염기의 품질 점수를 추출하고, numpy 모듈을 사용하여 평균, 중위값, 상위 25%, 하위 25%를 계산한다. 이와 같이 품질 점수의 통계치를 통해 데이터의 신뢰성을 평가할 수 있다.
'bioinformatics' 카테고리의 다른 글
| 한국에서의 DTC 유전자검사: 검사 항목, 방법, 규제 및 글로벌 차이점 (1) | 2024.11.02 |
|---|---|
| 멀티오믹스 분석 주제와 활용할 수 있는 공공 데이터베이스 (3) | 2024.10.29 |
| Argo workflow를 사용한 파이프라인 관리 (0) | 2024.09.22 |
| 연구실 정보 관리 시스템 (LIMS)의 사용 (0) | 2024.08.26 |
| NIPT 분석 (0) | 2021.06.01 |